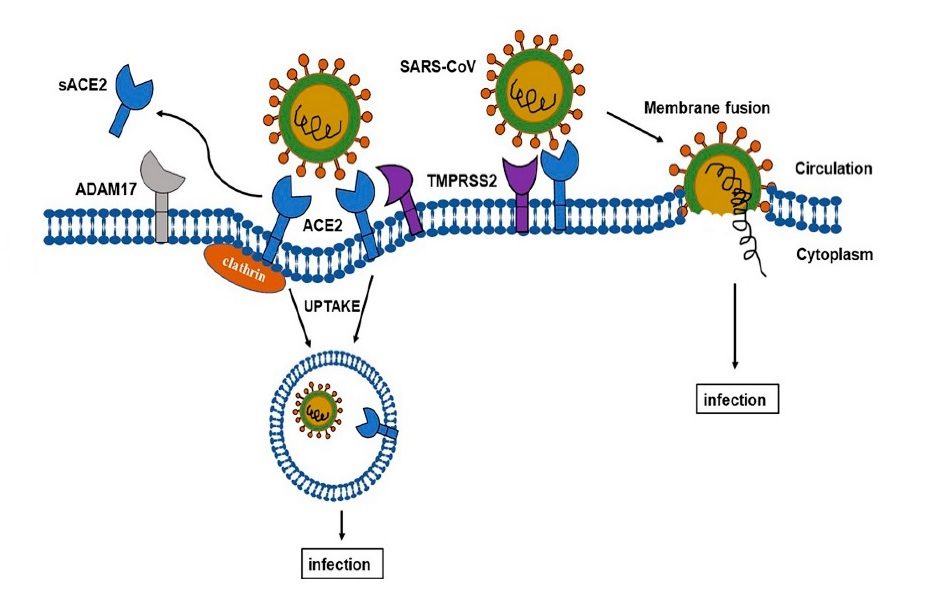
A ACE2 é uma proteína transmembrana expressa na superfície de diversas células do corpo, como o epitélio do sistema respiratório. Certamente, vários estudos já demonstraram a relação entre a proteína ACE2 com os mecanismos de entrada de alguns coronavírus, como o HCoV-NL63, o SARS-CoV e o novo SARS-CoV-2 (causador da COVID-19).
Guilherme Damasio, mestre em Microbiologia, Parasitologia e Patologia (com dissertação sobre Vírus Respiratórios e Síndrome Respiratória Aguda Grave – SRAG) e integrante do Grupo de Suporte Científico da Biometrix Diagnóstica, explica como essa correlação pode ser útil para o avanço nas pesquisas e combate à COVID-19.
Qual a função do gene ACE2 no organismo?
A princípio, o gene é responsável pela expressão da proteína de mesmo nome, ACE2 (da sigla em inglês: angiotensin-converting enzyme 2). Essa proteína está expressa na superfície das células e nada mais é do que um homólogo da já conhecida ACE (em português: ECA) – responsável pela regulação da pressão arterial dentro do Sistema Renina-Angiotensina.
Nesse sentido, a variante ACE2, descoberta no ano 2000, é muito semelhante em estrutura (cerca de 42%) mas faz o papel inverso da ACE. Enquanto a última faz vasoconstrição e consequente aumento da pressão arterial, a ACE2 promove a vasodilatação e diminui a mesma. Dessa forma é feita a regulação do Sistema Renina-Angiotensina.
Por que pessoas com doenças crônicas tem mais chances de serem infectadas pelo novo coronavírus (SARS-CoV-2)?
Isso se deve pelo fato de o gene ACE2 geralmente ser mais expresso em pacientes com doenças crônicas como hipertensão, diabetes e outras doenças cardiovasculares, o que as torna mais suscetíveis a infecção pelo novo coronavírus. Além disso, o uso de medicamentos inibidores da enzima conversora de angiotensina (iECA) e dos bloqueadores dos receptores de angiotensina (BRA), comumente usados por pacientes com diabetes e hipertensão, pode resultar em um aumento ainda maior da expressão de ACE2 devido a um mecanismo compensatório (upregulation).
Sobretudo, como a entrada do SARS-CoV-2 na célula é feita principalmente por meio da ligação da Proteína Spike do vírus com o receptor ACE2, o aumento da expressão dessa molécula na superfície das células desses pacientes pode aumentar a chance de infecção e até mesmo influenciar na gravidade da doença.
No entanto, é sempre importante lembrar que cada caso deve ser avaliado de forma individual pelo médico, principalmente com relação à medicação desses pacientes com comorbidades e nunca se deve automedicar nem parar qualquer tratamento sem antes consultar um especialista.
Um estudo recém publicado na plataforma medRxiv mostra que existem diversas variantes do gene ACE2. Qual a relevância dessas mutações no gene ACE2 tendo em vista a forma de infecção do SARS-CoV-2?
Esse estudo ainda é muito teórico, assim sendo, foram feitos apenas modelos preditivos de como seria essa interação. No entanto, os resultados obtidos foram muito promissores. Foi observado que o gene ACE2 apresenta muitos polimorfismos, alguns deles inclusive associados a doenças como diabetes e hipertensão.
Dessa forma, esses polimorfismos são causados por mutações no DNA, o que resultou em diferentes variantes do gene e consequentemente da proteína expressa. As mutações não chegam a interferir no funcionamento da enzima, pois ficam fora do sítio catalítico da mesma, mas alteram a estrutura tridimensional da molécula em algumas regiões. Sobretudo, essas alterações estruturais podem dificultar a ligação do vírus e sua entrada nas células, conforme demonstrado no artigo.
Isso também pode explicar por que algumas pessoas tem sintomas mais leves que outras da COVID-19?
A princípio, tudo indica que sim. Pelos resultados apresentados no estudo acima, algumas mutações no gene ACE2 (e consequente alteração estrutural da proteína) podem conferir teoricamente um fator de proteção contra a infecção pelo SARS-CoV-2, resultando em melhor prognóstico com relação à COVID-19. Aliás, isso pelo menos explicaria em partes porque as pessoas reagem de forma tão diferente à doença, em diferentes nacionalidades e etnias.
Para corroborar com os dados, são citados no artigo dados já amplamente conhecidos na literatura sobre o fato de indivíduos possuírem diferentes mutações genéticas que podem interferir na susceptibilidade ou resistência a algumas variantes do HIV.
É possível inibir a expressão de ACE2 no organismo? Que riscos poderia trazer ao paciente?
Já foi discutido sobre se essa possibilidade ajudaria a combater a infecção. Por outro lado, apesar de a ACE2 ser a porta de entrada do vírus nas células, foi mostrado em outros artigos que a ação da ACE2 tem efeito protetor contra danos pulmonares causados por infecções virais. Consequentemente, os estudos mostraram que a diminuição nos níveis dessa proteína pode causar ainda mais danos aos pulmões de quem está infectado. Pois essa diminuição na expressão de ACE2 pode levar ao aumento da permeabilidade vascular, do edema pulmonar e do acúmulo de neutrófilos, além da piora na função pulmonar.
Entendimento e evolução da doença
Sobretudo, é essencial entender que ainda estamos na fase de aprendizagem sobre esse novo vírus e que a todo momento são feitas publicações científicas com novas informações, que mudam tudo que se sabia até o momento. Assim sendo, não podemos esquecer também que não é somente o receptor ACE2 que está envolvido na entrada do vírus nas células.
De certo, a molécula recém descrita TMPRSS2 também é fundamental para o processamento da proteína spike para que ocorra essa entrada. Isso abre mais novas possibilidades de alvos para tratamentos e entendimento da evolução da doença.
Referências:
“Gene: ACE2, angiotensin I converting enzyme 2”. National Center for Biotechnology Information (NCBI). U.S. National Library of Medicine. 2020-02-28.
Donoghue M, Hsieh F, Baronas E, Godbout K, Gosselin M, Stagliano N, Donovan M, Woolf B, Robison K, Jeyaseelan R, Breitbart RE, and Acton S (1 Sep 2000). “A Novel Angiotensin-Converting Enzyme–Related Carboxypeptidase (ACE2) Converts Angiotensin I to Angiotensin 1-9”. Circulation Research. 87 (5): e1–e9. doi:10.1161/01.RES.87.5.e1. PMID 10969042.
Keidar S, Kaplan M, Gamliel-Lazarovich A (February 2007). “ACE2 of the heart: From angiotensin I to angiotensin (1-7)”. Cardiovascular Research. 73 (3): 463–9. doi:10.1016/j.cardiores.2006.09.006. PMID 17049503.
Lambert DW, Yarski M, Warner FJ, Thornhill P, Parkin ET, Smith AI, et al. (August 2005). “Tumor necrosis factor-alpha convertase (ADAM17) mediates regulated ectodomain shedding of the severe-acute respiratory syndrome-coronavirus (SARS-CoV) receptor, angiotensin-converting enzyme-2 (ACE2)”. The Journal of Biological Chemistry. 280 (34): 30113–9. doi:10.1074/jbc.M505111200. PMID 15983030.
Fehr AR, Perlman S (2015). “Coronaviruses: an overview of their replication and pathogenesis”. Methods in Molecular Biology. Springer New York. 1282: 1–23. doi:10.1007/978-1-4939-2438-7_1. ISBN 978-1-4939-2437-0. PMC 4369385. PMID 25720466.
Diaz JH (March 2020). “Hypothesis: angiotensin-converting enzyme inhibitors and angiotensin receptor blockers may increase the risk of severe COVID-19”. Journal of Travel Medicine. doi:10.1093/jtm/taaa041. PMID 32186711.
Hoffmann M, Kleine-Weber H, Schroeder S, Kruger N, et al. “SARS-CoV-2 Cell Entry Depends on ACE2 and TMPRSS2 and Is Blocked by a Clinically Proven Protease Inhibitor”. Cell. 2020;181:1-10. doi: 10.1016/j.cell.2020.02.052. PMID: 32142651
Imai Y, Kuba K, Penninger JM (May 2008). “The discovery of angiotensin-converting enzyme 2 and its role in acute lung injury in mice”. Experimental Physiology. 93 (5): 543–8. doi:10.1113/expphysiol.2007.040048. PMID 18448662.
Jia H (September 2016). “Pulmonary Angiotensin-Converting Enzyme 2 (ACE2) and Inflammatory Lung Disease”. Shock. Augusta, Ga. 46 (3): 239–48. doi:10.1097/SHK.0000000000000633. PMID 27082314.
Artigo citado na matéria (Plataforma medRxiv):
Dr. Mushtaq Hussain, Dr. Nusrat Jabeen, Fozia Raza, Sanya Shabbir, Ayesha Ashraf Baig, Anusha Amanullah and Basma Aziz. “Structural Variations in Human ACE2 may Influence its Binding with SARS-CoV-2 Spike Protein”. Journal of Medical Virology. https://doi.org/10.1002/jmv.25832.