Carlos Brogna, Simone Cristoni, Juliano Marinho, Luís Montano, Valentina Viduto, Mark Fabrowski, Gennaro Lettieri, Marina Piscopo
Resumo
Propósito
A pandemia de SARS-CoV-2 levou ao desenvolvimento e uso de vacinas de última geração. Entre elas, as vacinas baseadas em mRNA consistem em soluções injetáveis de mRNA que codificam uma Spike recombinante, que é distinguível da proteína do tipo selvagem devido a variações específicas de aminoácidos introduzidas para manter a proteína em um estado pré-fundido. Este trabalho apresenta uma abordagem proteômica para revelar a presença da proteína Spike recombinante em indivíduos vacinados, independentemente do título de anticorpos.
Desenho experimental
O exame de espectrometria de massa de amostras biológicas foi usado para detectar a presença de fragmentos específicos da proteína Spike recombinante em indivíduos que receberam vacinas baseadas em mRNA.
Resultados
O fragmento específico PP-Spike foi encontrado em 50% das amostras biológicas analisadas, e sua presença foi independente do título de anticorpos IgG do SARS-CoV-2. O tempo mínimo e máximo em que o PP-Spike foi detectado após a vacinação foi de 69 e 187 dias, respectivamente.
Conclusões e relevância clínica
O método apresentado permite avaliar a meia-vida da molécula de proteína Spike “PP” e considerar os riscos ou benefícios em continuar a administrar doses de reforço adicionais da vacina de mRNA do SARS-CoV-2. Esta abordagem é de suporte valioso para complementar o monitoramento do nível de anticorpos e representa a primeira detecção proteômica de Spike recombinante em indivíduos vacinados.
1 INTRODUÇÃO
Síndrome Respiratória Aguda Grave coronavírus 2 (SARS-CoV-2) é o coronavírus causador da doença respiratória COVID-19 (doença do coronavírus 2019), responsável pela pandemia em curso que mantém o mundo inteiro sob controle. Pesquisadores ao redor do mundo durante esses anos de pandemia estudaram esse vírus, tentando entender seu mecanismo de ação. O genoma de RNA do SARS-CoV-2 consiste em cerca de 30.000 nucleotídeos e contém 11 genes codificadores principais. Do ponto de vista estrutural, o SARS-CoV-2 é caracterizado por um grande número de proteínas Spike (S) glicosiladas cobrindo sua superfície e facilitando a ligação ao receptor da enzima conversora de angiotensina 2 (ACE2) da célula hospedeira, mediando a entrada na célula viral. A proteína Spike (S) é uma das quatro principais proteínas do SARS-CoV-2. Ela permite o reconhecimento do receptor da célula hospedeira e a subsequente entrada na célula hospedeira. Consiste na subunidade S1 distal, que é útil para o reconhecimento, e na subunidade S2 proximal, que é essencial para a fusão com a membrana da célula hospedeira. Durante o desenvolvimento das duas vacinas baseadas em mRNA mais amplamente utilizadas, Pfizer-BioNTech (BNT162b2- Comirnaty) e Moderna (mRNA-1273), todas as bases de nitrogênio de uridina foram substituídas por metil pseudouridina (m1Ψ) — uma base de nitrogênio menos imunogênica. Ao mesmo tempo, mutações foram feitas dentro dos 4284 nucleotídeos que constituem a proteína Spike, nas posições K986P e V987P para tornar a proteína produzida após a leitura ribossômica estável na forma de pré-fusão para estimular o aumento da produção de anticorpos humanos (Figura 1A). A proteína S do SARS-CoV-2 é altamente conservada entre todos os coronavírus humanos (HCoV) e está envolvida no reconhecimento do receptor, na fixação viral e na entrada nas células hospedeiras. Por esse motivo, representa, para uma parte dos cientistas, um dos alvos mais importantes para o desenvolvimento de vacinas e abordagens terapêuticas contra a COVID-19. Entre as vacinas contra a COVID-19 desenvolvidas e testadas, aquelas que mostraram os resultados mais promissores na prevenção da infecção pela COVID-19 são uma nova classe de produtos vacinais compostos por filamentos de ácido ribonucleico mensageiro (mRNA) encapsulados em nanopartículas lipídicas (LNPs). Duas delas receberam “autorização de uso emergencial” (da Federal and Drug Administration) e “aprovação condicional” (da Agência Europeia de Medicamentos). Ambas consistem em um mRNA recombinante a ser inoculado como vacina, que codifica uma proteína Spike recombinante do SARS-CoV-2. Embora os mRNAs sejam diferentes, ambos codificam a mesma proteína Spike recombinante (aqui chamada de PP-Spike). Isso difere do natural (wt-Spike, Figura 1B) por uma dupla mudança de aminoácido na posição 986 e 987 (K986P e V987P, ou seja, os aminoácidos lisina e valina são ambos substituídos por dois aminoácidos prolina), a fim de estabilizar a conformação Spike em um estado de pré-fusão inativo (Figura 1A). A variação de aminoácido dupla introduzida abole um sítio de digestão tríptica. Como resultado, é possível distinguir, por digestão tríptica, seguida por análise de espectrometria de massa, proteínas Spike sintéticas originadas da tradução das vacinas de mRNA da Spike natural circulando em fluidos biológicos. Aqui apresentamos uma abordagem metodológica que pode detectar especificamente a presença de PP-Spike em fluidos biológicos de organismos humanos e animais, como sangue, urina, saliva e fluidos de lavagem broncoalveolar.
Importância do estudo
Embora a pandemia da COVID-19 tenha deixado o mundo inteiro de joelhos, ela também permitiu que muitos cientistas desenvolvessem ideias e soluções contra vírus. Isso inclui vacinas de mRNA, que, devido à sua versatilidade na produção, podem representar um novo padrão de vacinação. No entanto, é dever do cientista não negligenciar os controles. Aqui reside a importância de monitorar a proteína Spike “PP” induzida pela vacina após um período de tempo após a vacinação em amostras biológicas humanas. O método apresentado permite avaliar a meia-vida da molécula de proteína Spike “PP” e considerar os riscos ou benefícios em continuar com doses de reforço adicionais da vacina de mRNA SARS-CoV-2.
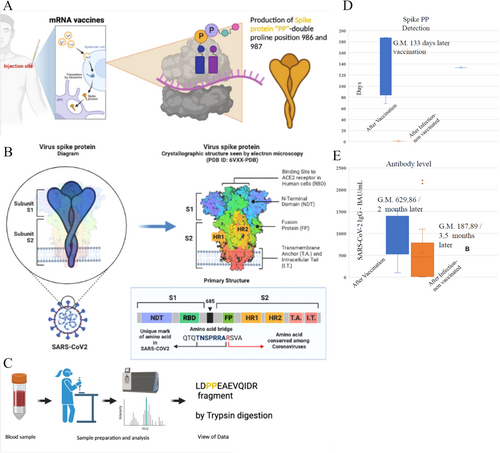
O grupo de estudo, do sul da Itália, foi de 40 indivíduos, 20 foram vacinados com o ciclo completo da vacina de mRNA em abril de 2022, fazendo parte do setor de saúde, e 20 não foram vacinados com negatividade para COVID-19 no teste nasofaríngeo e sem título de nenhum anticorpo. Outras 20 pessoas não vacinadas foram adicionadas que foram positivas para COVID-19. O fragmento PP-Spike específico foi encontrado em 50% das amostras biológicas analisadas (Figuras 1C–E e 2). Essa presença foi independente do título de anticorpos IgG do SARS-CoV-2. Os títulos de anticorpos tiveram uma média geométrica de 629,86 BAU/mL (Figura 1E). O tempo mínimo em que o PP-Spike foi detectado foi 69 dias após a vacinação, enquanto o tempo máximo foi 187 dias. Todos os controles (amostras de indivíduos não vacinados) foram negativos. O grupo de controle (20 pessoas não vacinadas) também foi testado após contrair COVID-19 e apresentou resultado negativo para PP-spike.
Alguns estudos observaram a presença da proteína Spike da vacina imediatamente após a injeção.
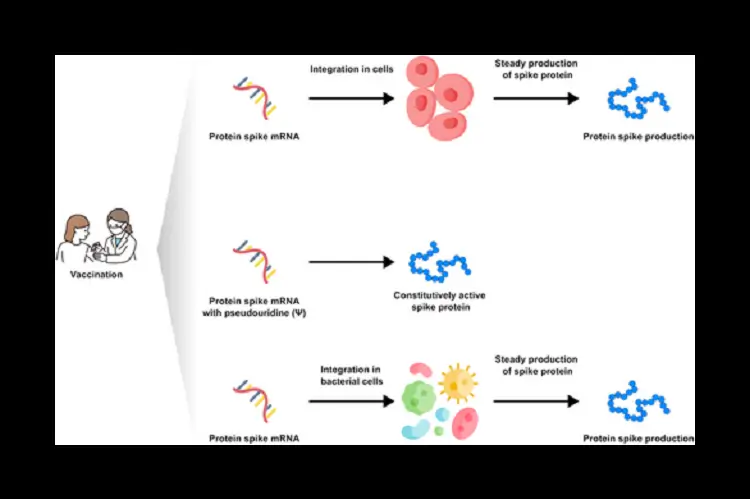
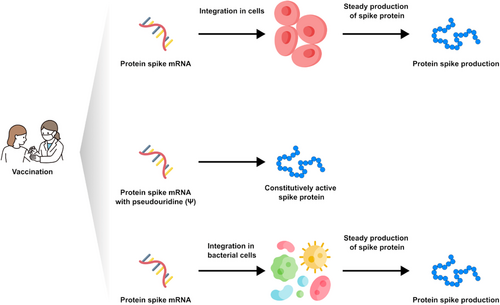
De acordo com os autores e em geral, as moléculas de nanopartículas de RNA mensageiro da vacina devem ser captadas pelas células imunes nos gânglios linfáticos após a injeção no músculo. Recentemente, outros autores isolaram sequências de RNA mensageiro da vacina do plasma periférico após 28 dias após a injeção. A questão de se o RNA da vacina pode ou não ser integrado ao linfócito ou outras células do corpo é muito debatida. No entanto, a observação da proteína produzida, conforme apresentada neste manuscrito, vai além do aspecto puramente cognitivo e define um método para verificar não apenas a persistência do RNA da vacina, mas a quantificação do produto, ou seja, a proteína que deve induzir a produção de anticorpos, a fim de verificar a meia-vida correta e uma possível necessidade de atualização do status da vacina. Usando o exame de espectrometria de massa de amostras biológicas, detectamos a presença de fragmentos específicos da proteína Spike recombinante em cerca de 50% dos indivíduos que receberam vacinas baseadas em mRNA. Em alguns casos, encontramos o marcador PP-Spike em indivíduos vacinados mais de 30 dias após a vacina, indicando que é possível detectar a proteína “Spike” da vacina mesmo algum tempo após a vacinação e em qualquer tecido orgânico (dados em preparação). Com base nos resultados obtidos, hipóteses podem ser feitas para possíveis mecanismos moleculares de persistência da “Spike PP”. Em particular, três hipóteses são possíveis e são mostradas na Figura 3.
- É possível que o mRNA seja integrado ou transcrito novamente em algumas células.
- É possível que pseudo-uridinas em uma posição de sequência específica, como descrito no artigo, induzam a formação de uma proteína spike que é sempre constitutivamente ativa. Mas parece muito remoto como hipótese.
- É possível que a nanopartícula contendo mRNA seja captada por bactérias normalmente presentes no nível basal no sangue. De fato, a existência de microbiota sanguínea em indivíduos clinicamente saudáveis foi comprovada durante os últimos 50 anos. De fato, evidências indiretas por análises radiométricas sugeriram a existência de formas microbianas vivas em eritrócitos. Além disso, a observação do marcador PP-Spike em indivíduos vacinados mais de 30 dias após a vacina em cerca de 50% dos indivíduos também pode ser explicada pela ampla biodiversidade de microbiota eucariótica e procariótica identificada no sangue por tecnologias de sequenciamento de última geração.
Concluindo, a possibilidade de detectar a presença de fragmentos específicos da proteína Spike recombinante abre novos cenários para o monitoramento da presença e da meia-vida da proteína Spike vacinal em indivíduos vacinados. As hipóteses avançadas precisam de mais e maiores estudos. Atualmente, essas observações iniciais se limitam apenas a avaliar a presença da proteína vacinal com o objetivo de querer auxiliar na decisão do indivíduo de administrar doses de reforço ou temporizar.
2 DADOS ASSOCIADOS
Análise estatística: teste t com © 2023 GraphPad Software foi realizado entre os dois grupos, vacinados e não vacinados para a presença do peptídeo de spike chamado PP. ” Valor de p e significância estatística: O valor de P bicaudal é igual a 0,0010. Intervalo de confiança: A média do grupo um menos o grupo dois é igual a 0,40, intervalo de confiança de 95% dessa diferença: 0,17–0,63. Valores intermediários usados nos cálculos: t = 35.590, df = 38, erro padrão da diferença = 0,112. A diferença no título de anticorpos entre os dois grupos não é significativa.
3 PROCEDIMENTOS EXPERIMENTAIS
De acordo com os regulamentos territoriais aplicáveis, relatos de casos, séries de casos e estudos retrospectivos observacionais não exigem aprovação do comitê de ética. O consentimento informado expresso por todos os participantes, no sul da Itália, foi obtido. Vinte amostras biológicas humanas foram coletadas de indivíduos vacinados com consentimento expresso, livre e informado para coleta e uso. A média geométrica do título de seus anticorpos versus proteína spike foi de 629,86 BAU/mL após 60 dias da vacinação. Além disso, 20 amostras biológicas humanas foram coletadas de indivíduos não vacinados (grupo controle, negativo) com consentimento expresso, livre e informado para coleta e uso. Os candidatos não haviam sido submetidos à COVID-19 anteriormente e foram negativos para testes moleculares por swab nasofaríngeo, e o título de anticorpos foi negativo. Outras 20 amostras biológicas humanas de pacientes não vacinados, que estavam doentes com COVID-19, foram obtidas (a média geométrica do título de anticorpos versus proteína spike foi de 187,89 BAU/mL após 105 dias da doença).
3.1 Pré-analítico
A coleta de amostra de sangue seco pré-clínica foi realizada em conformidade com a literatura científica: a técnica de Ponto de Sangue Seco (DBS) é comum para a coleta de pequenas quantidades de sangue, especialmente de recém-nascidos para aplicação em triagem. Este método foi publicado pela primeira vez por Robert Guthrie no início da década de 1960 e é baseado na coleta de uma pequena gota de sangue em um papel de filtro em vez de coletar alguns mililitros de sangue em um frasco. Com a técnica DBS, a coleta de amostras é fácil e rápida, sem a necessidade de profissionais treinados. No entanto, as pequenas quantidades de sangue coletadas (∼20–40 µL) tornam esta abordagem mais desafiadora analiticamente em termos de preparação e sensibilidade da amostra em comparação com a coleta de sangue tradicional. As gotas de sangue foram coletadas em modo estéril. Para preservar a estrutura da proteína, o filtro de sangue seco foi mantido em condições de -20 °C até o processamento.
3.2 Justificativa
A tripsina é uma enzima pertencente à classe das hidrolases capaz de reduzir proteínas a polipeptídeos menores por cortes proteolíticos com especificidade para arginina (R) e lisina (K). A proteína Spike sintética e a proteína Spike natural podem assim ser distinguidas, pois produzem diferentes produtos de digestão tríptica:
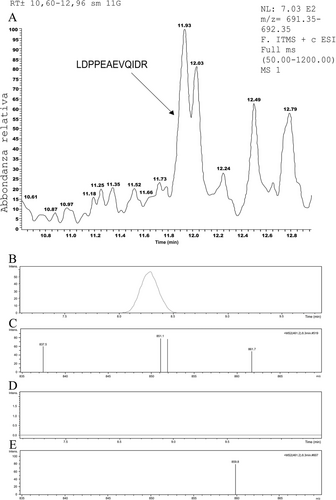
- – Quando digerido pela tripsina, o PP-Spike codificado pelo mRNA da vacina produz um fragmento LDPPEAEVQIDR (marcador PP-Spike) (Figura 2C).
- – A proteína SAR-CoV-2 do tipo selvagem, quando digerida pela tripsina, produz dois fragmentos menores, nomeadamente LDK + VEAEVQIDR.
Mais de 6.600.000 genomas do SARS-CoV-2 foram sequenciados e parece que nenhum deles tem mutações K986P e V987P, incluindo a variante Ômicron.
Tampões utilizados: Água bidestilada (VWR); bicarbonato de amônio (NH4 HCO3) (Sigma Aldrich). Reagentes: Tripsina da Promega.
Preparação do reagente: Solução de tripsina 25 ng/µL.
Procedimento de preparação: Ressuspenda, em um frasco, 20 µg de tripsina sólida em 800 µL de solução de NH4 HCO3 50 mM. Agite o frasco no vórtice até que a tripsina esteja completamente dissolvida.
O peptídeo padrão correspondente ao fragmento LDPPEAEVQIDR (Sigma Aldrich, Reino Unido) foi diluído para uma concentração de 10 ng/mL em água bidestilada. Dez microlitros foram injetados no aparelho LC-MS para fins de controle. O peptídeo detectado no plasma foi caracterizado com base nos regulamentos da UE.
3.3 Digestão enzimática
O procedimento de digestão enzimática de gotas de sangue é realizado sob uma capela de exaustão para minimizar a exposição do operador a qualquer forma de risco biológico químico. O método a seguir foi usado para cada amostra de sangue. Coloque 2 µL de sangue capilar em um tubo Eppendorf rotulado; adicione 40 µL de tripsina (Promega, Itália) solubilizada em uma solução de 50 mmoL de NH4 HCO3 à amostra de sangue; agite por 30 s; teste o pH, que deve estar entre 7 e 8; transfira o tubo Eppendorf para o termobloco; incube por 2 dias a 37 °C; adicione 40 µL de NH4 HCO3 50 mmoL. Colete 40 µL do sobrenadante obtido após centrifugação a 13000 G por 10 min e transfira para um tubo Eppendorf. Adicione 2 µL de ácido fórmico puro e transfira a solução para um frasco de injeção. O frasco é inserido no amostrador automático acoplado ao espectrômetro de massas e 5 µL são injetados na coluna cromatográfica.
3.4 Instrumentação LC–SACI–MS
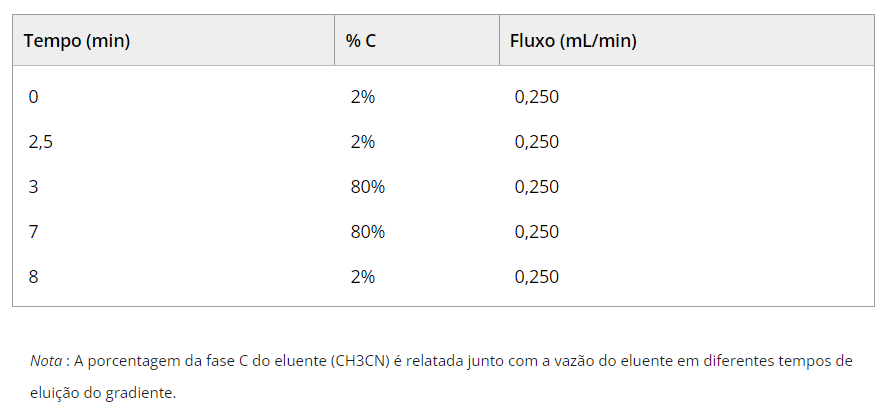
A análise foi realizada com um Surveyor MS HPLC (Thermofisher, EUA). A coluna utilizada foi uma Kinetex 50 × 4,6 mm 2,6 µm. A análise foi realizada usando um gradiente de duas fases: Fase A (H 2 O + 0,2 % ácido fórmico (HCOOH)) e Fase C (CH 3 CN) (Tabela 1). O gradiente cromatográfico utilizado é mostrado na Tabela 1. O volume da amostra injetada é de 5 µL. A fonte de ionização utilizada é um SACI-ESI. Um potencial de superfície de 0 V, uma pressão de gás nebulizador de 75 PSI e um fluxo de gás seco de 1,0 L/min foram utilizados. A temperatura do gás seco é de 320 °C.
TABELA 1. Condições cromatográficas de eluição do gradiente.
Fonte: https://pubmed.ncbi.nlm.nih.gov/37650258/