RESUMO
A doença de coronavírus 2019 (COVID-19), causada pelo coronavírus 2 da síndrome respiratória aguda grave (SARS-CoV-2), surgiu como uma pandemia e causou enormes danos à vida das pessoas e à economia de muitos países em todo o mundo. No entanto, os agentes terapêuticos contra o SARS-CoV-2 permanecem obscuros. O SARS-CoV-2 possui uma proteína spike (proteína S), e a clivagem da proteína S é essencial para a entrada viral. A nattoquinase é produzida por Bacillus subtilis var. nattoe é benéfico para a saúde humana. Neste estudo, examinamos o efeito da nattokinase na proteína S do SARS-CoV-2. Quando os lisados celulares transfectados com proteína S foram incubados com nattokinase, a proteína S foi degradada de maneira dependente da dose e do tempo. A análise de imunofluorescência mostrou que a proteína S na superfície da célula foi degradada quando nattokinase foi adicionada ao meio de cultura. Assim, nossas descobertas sugerem que a nattokinase exibe potencial para a inibição da infecção por SARS-CoV-2 por meio da degradação da proteína S.
1. INTRODUÇÃO
A doença de coronavírus 2019 (COVID-19), causada pelo coronavírus 2 da síndrome respiratória aguda grave (SARS-CoV-2), está se espalhando em todo o mundo. A pandemia de COVID-19 afetou mais de 437 milhões de pessoas e causou mais de 6,3 milhões de mortes (https://covid19.who.int/, acessado em 4 de julho de 2022). A entrada do SARS-CoV-2 nas células hospedeiras é mediada pela proteína spike transmembrana (proteína S), que forma homotrímeros que se estendem do envelope viral. A proteína S é processada e ativada por proteases celulares, incluindo proteína serina transmembrana 2 (TMPRSS2), catepsina e furina. Compreende duas subunidades funcionais, S1 e S2; a subunidade S1 do SARS-CoV-2 inicia a ligação do vírus ao receptor interagindo com a enzima conversora de angiotensina 2 (ACE2) do receptor da célula hospedeira humana, e a subunidade S2 participa da fusão viral com a célula-alvo, permitindo a entrada viral. O domínio de ligação ao receptor (RBD) na subunidade S1 é responsável pela ligação ao ACE2. A clivagem da proteína S ocorre no limite entre as subunidades S1 e S2.
Atualmente, muitos países estão envolvidos no desenvolvimento de vacinas para proteger contra a infecção por SARS-CoV-2, portanto, o número de infecções por SARS-CoV-2 diminuiu. No entanto, numerosas variantes de SARS-CoV-2, incluindo cepas com epítopos-alvo de vacina mutantes, foram relatadas. A vacinação pode não proteger completamente contra a infecção por SARS-CoV-2 porque o número de pacientes com COVID-19 está aumentando após a vacinação. Portanto, é importante desenvolver novos tratamentos para infecções por SARS-CoV-2.
Natto é um popular alimento tradicional japonês feito de soja fermentada por Bacillus subtilis var. natto. A nattoquinase é encontrada no natto e é uma das mais importantes enzimas extracelulares produzidas por B. subtilis var. natto. Nattokinase consiste em 275 aminoácidos e é de aproximadamente 28 kDa. A nattoquinase inativa o inibidor-1 do ativador do plasminogênio e aumenta a fibrinólise. Também diminui os níveis plasmáticos de fibrinogênio, fator VII, citocinas e fator VIII. A nattoquinase tem a maior potência de dissolução de coágulos entre os anticoagulantes naturalmente conhecidos. Um ensaio clínico demonstrou que o consumo oral de nattokinase não foi associado a nenhum efeito adverso. Assim, a nattokinase é agora considerada uma enzima eficiente, segura e econômica que tem atraído a atenção central em estudos de drogas trombolíticas. Além disso, a nattoquinase é utilizada no tratamento de alguns tumores.
Um estudo recente revelou que o extrato de natto inibe a infecção por herpesvírus bovino 1 (BHV-1) e SARS-CoV-2. Esses resultados indicam que a protease do extrato de natto pode ser eficaz contra a infecção por SARS-CoV-2. Neste estudo, objetivamos investigar se a inibição da infecção por SARS-CoV-2 pelo extrato de natto é causada pela nattokinase derivada de B. subtilis var. natto.
2. RESULTADOS E DISCUSSÃO
2.1. Efeitos degradativos da nattoquinase na proteína Spike do SARS-CoV-2 in vitro
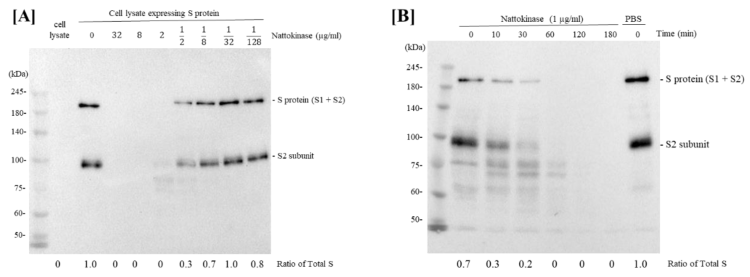
Primeiro investigamos se a nattokinase no extrato de natto poderia degradar a proteína SARS-CoV-2 S. A proteína S do SARS-CoV-2 desempenha um papel importante no receptor ACE2 da célula hospedeira durante os estágios iniciais da infecção. Depois de misturar o lisado celular de expressão da proteína S com uma série de diluição de 4 vezes de nattokinase (32 µg/mL, 8 µg/mL, 2 µg/mL, 500 ng/mL, 125 ng/mL, 31,25 ng/mL e 7,8125 ng/mL), Western blotting foi realizado. O comprimento total da proteína S (subunidades S1 e S2) e a subunidade S2 apareceram como bandas quando o lisado celular de expressão da proteína S foi incubado com D-PBS em concentrações de nattoquinase de 500 ng/mL, 125 ng/mL, 31,25 ng/mL e 7,8125 ng/mL (Figura 1 A). Em seguida, examinamos se a nattokinase degrada a proteína S de maneira dependente do tempo. O lisado foi então incubado com 1 µg/mL de nattoquinase por 10 a 180 min. A proteína S do SARS-CoV-2 foi degradada pela nattokinase após 60 a 180 min de incubação, mas não após 10 e 30 min de incubação (Figura 1 B). Assim, a nattokinase degradou a proteína S de maneira dependente da dose e do tempo.
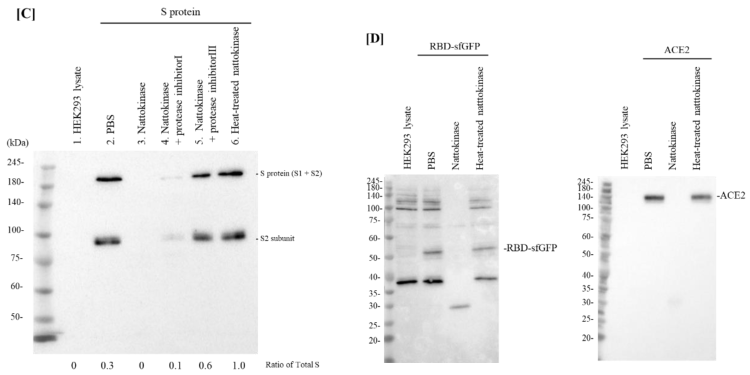
Figura 1. (A) Efeitos degradativos da natoquinase de maneira dependente da dose. Nattokinase diluída em série (32 µg/mL, 8 µg/mL, 2 µg/mL, 500 ng/mL, 125 ng/mL, 31,25 ng/mL e 7,8125 ng/mL) foram misturados com lisado de células de expressão de proteína S e incubados. Comprimento total da proteína S (subunidades S1 e S2) e subunidade S2 foram detectados como bandas superiores e inferiores, respectivamente. A proporção de S total foi indicada como a quantidade relativa de proteína S (proteína S + proteína S2). (B) Efeitos degradativos da nattokinase de maneira dependente do tempo. O lisado de células de expressão da proteína S foi incubado com 1 µg/mL de nattoquinase por 0, 10, 30, 60, 120 e 180 min. (C) Efeitos do tratamento térmico ou inibidores de protease. Pista 1: lisado de HEK293; pista 2: lisado HEK293 (proteína S); pista 3: HEK293 (proteína S) + natoquinase (5 µg/mL); pista 4: HEK293 (proteína S) + natoquinase (5 µg/mL) + inibidor de protease I; pista 5: HEK293 (proteína S) + natoquinase (5 µg/mL) + inibidor de protease III; pista 6: HEK293 (proteína S) + natoquinase tratada termicamente (5 µg/mL). (D) Efeito degradativo no RBD da proteína S e ACE2. RBD de proteína S e plasmídeos codificadores de ACE2 foram transfectados com células HEK293, respectivamente. Lisados celulares foram incubados com nattokinase (7,5 µg/mL) e nattokinase tratada termicamente (7,5 µg/mL) e Western blotting foi realizado.
Para confirmar se o efeito degradativo da nattokinase é devido à atividade enzimática, a nattokinase foi tratada com aquecimento ou um coquetel inibidor de protease. Quando a nattokinase foi aquecida a 100 °C por 5 min, o efeito degradativo da nattokinase foi perdido (Figura 1 C, faixa 6). Além disso, a perda das bandas da proteína S pela nattokinase foi bloqueada quando os inibidores de protease foram adicionados (Figura 1 C, pistas 4 e 5). Comparado com o coquetel inibidor de proteína I, o coquetel de proteína III, que consistia em AEBSF HCl (cloridrato de fluoreto de 4-(2-aminoetil) benzenossulfonil), aprotinina, que é um inibidor irreversível de serina protease, e leupeptina, que é uma cisteína-protease, claramente bloqueou a atividade da nattokinase. Nattokinase tem os mesmos aminoácidos conservados, Ser-His-Asp (Asp 32, His 64 e Ser 221), que são membros da família subtilisina de serina proteases. A estrutura cristalina da nattokinase é quase idêntica à da subtilisina E de B. subtilis DB104. Este resultado é consistente com o de um relatório anterior de que a nattokinase é uma serina protease. Também avaliamos os efeitos degradativos da nattokinase usando lisados celulares que expressam o RBD e o ACE2. Quando 7,5 µg/mL de nattokinase e lisado celular foram incubados, as bandas de RBD e ACE2 foram perdidas (Figura 1 D).
2.2. Efeitos degradativos da nattokinase na proteína Spike de SARS-CoV-2 na superfície celular transfectada
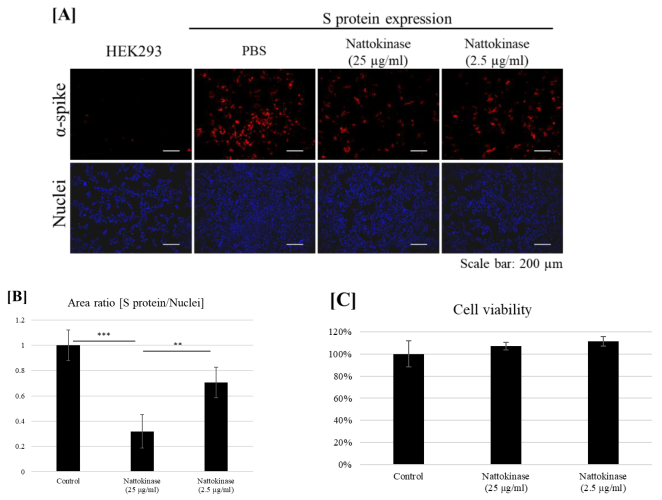
Em seguida, examinamos se a nattokinase degrada a proteína S na superfície celular transfectada. A proteína S foi transfectada com as células HEK293. As células transfectadas foram incubadas com nattokinase por 9 h. A proteína S na superfície celular foi detectada usando um anticorpo anti-proteína S sem permeabilização celular (Figura 2 A). A proteína S foi detectada nas células transfectadas. Quando as células transfectadas foram tratadas com nattokinase, a proteína S na superfície celular diminuiu. Quando as células foram tratadas com 25 µg/mL e 2,5 µg/mL de natoquinase, a proporção de área positiva para proteína S para área positiva para núcleo diminuiu em aproximadamente 0,3 e 0,7, respectivamente (Figura 2 B). O efeito degradativo da nattoquinase foi observado quando não havia citotoxicidade (Figura 2C). A análise de Western blotting mostrou que a quantidade de proteína S total não mudou entre os tratamentos com nattokinase e controle (Figura Suplementar; Figura S1). Esses resultados indicam que a nattoquinase degradaria a proteína S do SARS-CoV-2 na faixa de concentração não tóxica.
Figura 2. (A) Efeito degradativo da nattokinase na proteína S na superfície celular. Spike-pcDNA3.1 foi transfectado com células HEK293 e incubado por 9 h. Após a incubação, nattokinase (25 e 2,5 µg/mL) foram adicionados ao meio de cultura e incubados por 13 h. As células foram fixadas e a análise imunofluorescente foi realizada. A proteína S na superfície da célula foi corada com anticorpo de proteína anti-spike (Vermelho) e o núcleo foi corado com DAPI (Azul). (B) Razão entre a área da proteína S e a área positiva do núcleo. Três imagens por amostra foram capturadas e as áreas positivas de proteína S/núcleo foram calculadas. Os dados são mostrados como média + SD, e p-valor foi determinado por análise de variância (ANOVA) unidirecional com o teste post-hoc de Tukey usando o software R (R-3.3.3 para Windows) (** p < 0,01; *** p < 0,001). (C) A viabilidade celular foi avaliada pelo ensaio MTT. A nattoquinase indicada foi adicionada ao meio de cultura e incubada por 13 h; O ensaio MTT foi realizado.
Neste estudo, mostramos que a atividade protease da nattokinase contribui para a degradação da proteína S. A nattokinase tem um efeito degradante não apenas nas proteínas S, mas também na ACE2 nas células hospedeiras. A especificidade da protease da nattokinase seria baixa, porque GAPDH, uma proteína doméstica, também foi degradada simultaneamente na avaliação in vitro da nattokinase misturada com o lisado celular (Figura suplementar; Figura S2). Por outro lado, quando adicionado às células, não apresenta nenhum efeito sobre a viabilidade celular e espera-se que atue como um agente protetor na superfície celular. Uma análise mais aprofundada dos produtos de degradação da nattokinase usando espectrometria de massa é necessária para entender os efeitos da proteólise.
A nattokinase possui a potente atividade de degradação da proteína SARS-CoV-2 S e também demonstrou exercer efeitos anti-ateroscleróticos, hipolipemiantes, anti-hipertensivos, antitrombóticos, fibrinolíticos, neuroprotetores, antiplaquetários e anticoagulantes. Pacientes com hipertensão e comorbidades cardiovasculares podem facilmente ficar muito doentes com COVID-19. Devido ao surgimento de inúmeras variantes de SARS-CoV-2, incluindo cepas com epítopos-alvo de vacina mutantes, a vacinação por si só pode não proteger completamente contra a infecção por SARS-CoV-2. Os extratos de nattokinase e natto têm potencial para serem desenvolvidos como uma nova geração de medicamentos para a prevenção e tratamento do COVID-19.
3. MATERIAIS E MÉTODOS
3.1. Materiais
A nattokinase foi obtida da Contek Life Science Co., Ltd. (Taipei City, Taiwan). A atividade da nattokinase foi de 60.000 FU/g (FU, unidade de fibrinólise). Os coquetéis de inibidores de protease I e III foram adquiridos da FUJIFILM Wako Pure Chemical Corporation (Osaka, Japão). O plasmídeo de expressão (pcDNA3.1-SARS2-Spike C9 com tag no C-terminal, pcDNA3.1-hACE2 e pcDNA3-SARS-CoV-2-S-RBD-sfGFP) foi adquirido da Addgene (Watertown, MA, EUA). As células HEK293 (JCRB9068) foram obtidas do JCRB Cell Bank (Osaka, Japão).
3.2. Cultura Celular e Western Blotting
As células HEK293 foram cultivadas a uma densidade de 3,5 × 105 células/mL em DMEM suplementado com 10% de FBS, L-glutamina, 100 U/mL de penicilina e 100 μg/mL de estreptomicina durante a noite. As células foram transfectadas com cada plasmídeo (pcDNA3.1-SARS2-Spike, pcDNA3-SARS-CoV-2-S-RBD-sfGFP ou pcDNA3.1-hACE2) e incubadas por 22 h. Após a incubação, as células cultivadas foram raspadas e lavadas com solução salina tamponada com fosfato de Dulbecco gelada (D-PBS). A contagem de células foi realizada e o tampão xTractor (Takara Bio Inc., Shiga, Japão) foi adicionado ao precipitado celular. Os lisados celulares foram centrifugados a 1300 × gpor 10 min a 4 °C e o sobrenadante foi transferido para novos tubos e armazenado a -80 °C até o uso. A concentração de proteína foi determinada pelo ensaio de proteína de ácido bicinconínico (BCA) usando um kit de ensaio BCA (Takara). Dez microlitros de nattokinase e 10 µL de lisado celular (1 µ µg/mL) foram incubados a 37 °C por 1 h. Quando os efeitos dos inibidores de protease foram usados, os conjuntos de coquetéis de inibidores de protease I e III foram diluídos 10 vezes com D-PBS e uma solução de coquetel de inibidores de protease de 10 µL foi adicionada à mistura de nattokinase e lisado celular. Volumes iguais da mistura de reação foram carregados e o Western blotting foi realizado. Os anticorpos primários incluíam anticorpo monoclonal de camundongo anti-rodopsina (C9) (1D4) (Santa Cruz Biotechnology, Dallas, TX, EUA), anticorpo monoclonal de camundongo anti-GAPDH (FUJIFILM Wako), anticorpo monoclonal de camundongo anti-tag GFP (Proteintech, Rosemont, IL, EUA) e anticorpo anti-ACE2 (Proteintech). Os anticorpos secundários incluem anticorpo de cabra anti-rato conjugado com HRP (Proteintech).
3.3. Ensaio de imunofluorescência
As células HEK293 foram cultivadas a uma densidade de 3,5 × 105 células/mL em uma câmara de 8 poços em DMEM suplementado com 10% FBS, L-glutamina, 100 U/mL de penicilina e 100 μg/mL de estreptomicina. As células foram transfectadas com pcDNA3.1-SARS2-Spike e incubadas por 9 h. Após a incubação, as células foram tratadas com as amostras, incubadas por 13 h e fixadas em paraformaldeído a 4% por 30 min. Após incubação com anticorpo monoclonal SARS-CoV/SARS-CoV-2 spike (1A9) (GeneTex, CA, EUA) por 1 h, foi incubado com anticorpo anti-rato de cabra conjugado com Cy3 por 1 h. As lâminas foram coradas com DAPI Fluoromount-G e observadas usando um microscópio de fluorescência (BZ-X710, Keyence, Osaka, Japão). As áreas positivas para a proteína S e positivas para o núcleo foram calculadas usando o software de análise anexado BZ-X710 (BZ-X Analyzer). A viabilidade celular foi avaliada usando o ensaio de brometo de 3-(4,5-dimetiltiazol-2-il)-2,5-difeniltetrazólio (MTT). As células foram cultivadas em placas de cultura de 24 poços. Após incubação a 37 °C por 24 horas, as amostras foram adicionadas a cada poço e incubadas por mais 13 horas. As células foram suspensas em 500 μL de DMEM contendo 500 μg/mL de MTT. Após incubação por 3 h a 37 ° C, 500 μL de isopropanol contendo 4 mM de HCl foram adicionados para dissolver MTT formazan. A absorbância foi medida a 570 nm usando um leitor de microplacas.
4. CONCLUSÕES
Neste estudo, demonstramos que a nattokinase, uma serina protease, degrada a proteína S do SARS-CoV-2. Para investigar se a nattokinase contida no extrato de natto poderia inibir a infecção por SARS-CoV-2, analisamos a degradação da proteína S misturando o lisado celular de expressão da proteína S e a nattokinase de maneira dependente da dose e do tempo. O RBD da proteína S se liga à porção distal da membrana da proteína ACE2. Foi relatado que o extrato de Natto inibe a infecção por SARS-CoV-2 em células Vero E6 por meio da degradação de RBD. Demonstramos que a degradação da proteína S pela nattokinase foi bloqueada por tratamentos de calor ou inibidores de proteínas. Nossos dados sugerem que a atividade protease da nattokinase desempenha um papel crucial na degradação da proteína S. Tomados em conjunto, esses achados apoiam a noção de que a inibição da infecção por SARS-CoV-2 pelo extrato de natto foi devida à degradação da proteína S pela nattokinase. Assim, nossos dados indicaram que os extratos de nattokinase e natto têm efeitos potenciais na inibição da entrada da célula hospedeira SARS-CoV-2 por meio da degradação da proteína S.